single cell multiome 10X library 및 cellranger-arc 원리에 대해 공부중이다.
추가로 공부하는 내용이 있다면 계속 추가할 예정이다.
1. 멀티옴 ATAC 라이브러리 구성
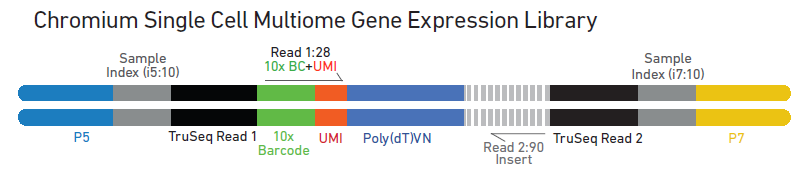
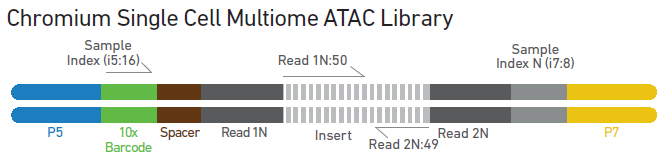
- P5, P7: Illumina amplification을 위한 서열
- 10X barcode: 세포를 구분하기 위한 cell barcode
- Spacer: ATAC oligo barcode(바코드)로, transposed DNA fragments가 결합(attachment)할 수 있는 바코드 (길이 8bp)
2. ATAC matrix 가공 과정
multiome FASTQ를 가공하여 peak-barcode 매트릭스를 만들기 위해서는 cellranger-arc count 코드 한 줄만 입력하면 되지만,
그 코드 한 줄에는 복잡한 가공과정이 포함되어 있다.
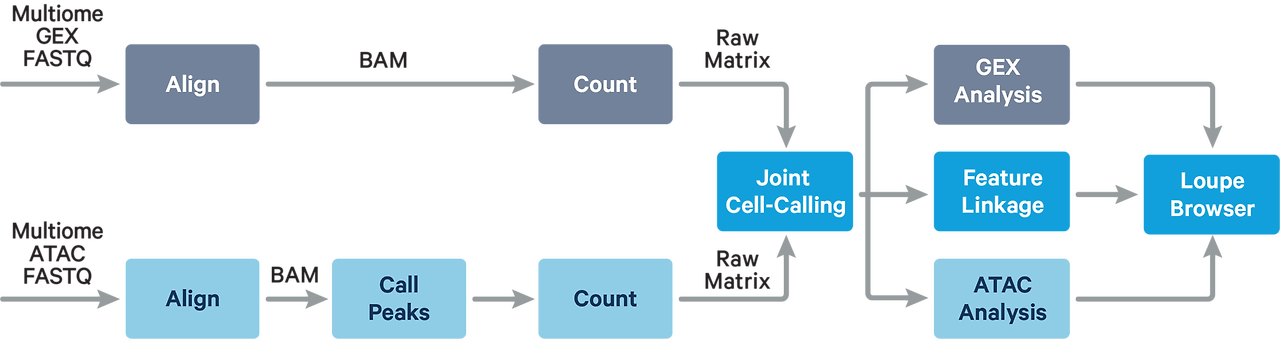
(Input) ATAC FASTQs ➡️ (Output) Peak-Barcode matrix
1. Barcode processing
: sequencing error에 의한 cell barcode 서열에 오류가 있는지 확인하는 과정으로, 10x의 valid barcode list와 비교하여 barcode의 valid를 평가함.
2. Read trimming
: 현재 기술로는 완벽히 원하는 서열만 읽어낼 수는 없고, read의 3' 말단에 primer(프라이머) 서열의 reverse complement가 포함되어 있을 수 있음. 이를 cutadapt 툴과 유사한 알고리즘을 통해 프라이머 서열을 인식하고 잘라냄(trimming).
3. Read alignmnent
: BWA-MEM 알고리즘을 이용
4. Duplicate marking
: PCR에 의한 duplication 탐지 및 제거하는 과정으로, fragments의 start, end position 및 cell barcode를 이용하여 duplicates를 찾음.
5. Peak calling
: fragments 중에는 nucleosome이 있는 영역이 포함되어 있음. 실제 TF가 접근할 수 있는 open chromatin 영역 즉, accessible regions을 찾기 위한 과정 ▶️ cellranger 결과 대신, macs2를 사용하길 권장
6. Peak-Barcode matrix
: Output
Reference
- https://support.10xgenomics.com/single-cell-multiome-atac-gex/software/pipelines/latest/glossary?src=website&lss=organic/direct/direct&cnm=wbr-2020-08-21-event-ra_g-p_scmage-apac-2020-08-21-event-ra_g-p_scmage-apac&cid=7011P000000oXe2
- https://genomebiology.biomedcentral.com/articles/10.1186/s13059-020-1929-3
Glossary of Terms -Software -Single Cell Multiome ATAC + Gene Exp. -Official 10x Genomics Support
10x GenomicsChromium Single Cell Multiome ATAC + Gene Exp. Cell Ranger ARC2.0, printed on 02/19/2024 Glossary of Terms Entries are ordered alphabetically. These definitions may be specific to the usage of these terms in the context of Chromium Next GEM Sin
support.10xgenomics.com
From reads to insight: a hitchhiker’s guide to ATAC-seq data analysis - Genome Biology
Assay of Transposase Accessible Chromatin sequencing (ATAC-seq) is widely used in studying chromatin biology, but a comprehensive review of the analysis tools has not been completed yet. Here, we discuss the major steps in ATAC-seq data analysis, including
genomebiology.biomedcentral.com
댓글